This is an old revision of the document!
Table of Contents
Graphical User Interface
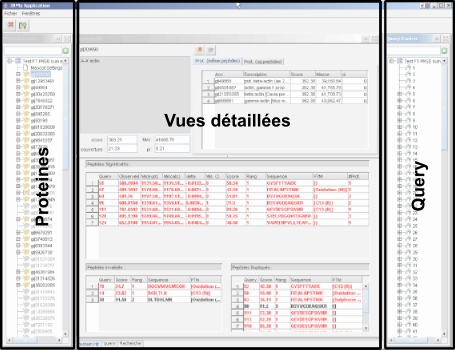
IRMa main window is splitted in three panels : the left one allow users to browse protein hits, the rigth one shows submitted queries and peptide sequences matching this queries. The center panel contains tabs representing Protein hit details, Search parameters, Query details and the list of identified proteins.
Browse protein hits
Center panel
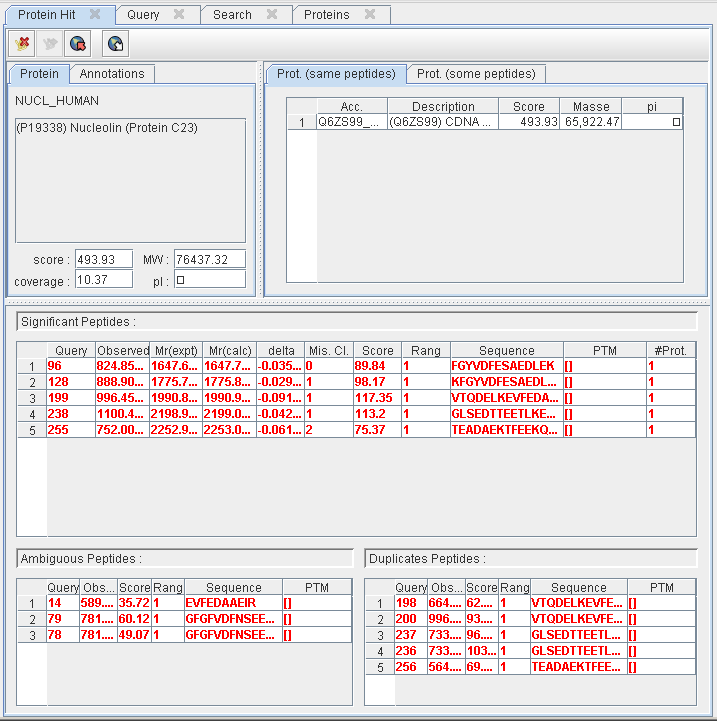
Protein Hit details
This pane displays the proteins and the set of peptides collapsed into the selected hit :
- The “main” protein details : description, score, mass, pi, coverage, etc.
- The collection of proteins matching the same set of peptides or a sub-set of this peptides.
- The set of peptides collapsed into this hit according to their category : significant, ambiguous or duplicated.
- Some additional informations associated to the “main” protein (emPAI, …)
At the top of this pane, a toolbar allow users to :
- delete the hit from the result if it does not contain any more significant PSM
- group the hit with another one if IRMa detect that the set of PSM classified as significants is a subset of PSM belonging to another hit.
The user can replace the main protein by another protein identified by the same set of PSM (right-clic on the protein you want to move as the new “main” protein).
Au niveau des peptides, il est possible d'effectuer les opérations suivantes sur cette vue :
- Obtenir la vue Mascot d'un peptide (peptide view) via une connexion au serveur Mascot. Pour ce faire, il faut sélectionner un peptide dans l'un des tableaux et presser la barre d'espace.
- Invalider un peptide en sélectionnant un ou plusieurs peptides dans le tableau des peptides significatifs et en pressant la touche A (pour Ambigu).
Query details
On retrouve ici, pour la query couramment sélectionnée, la liste des 10 peptides qui ont matchés. Pour chacun de ces peptides, il est possible de visualiser le spectre annoté et le tableau des ions fragments.
Search Parameters
Regroupe les informations sur la recherche Mascot : base utilisé, fichier soumis, taxonomie …
Proteins List
Liste de toutes les protéines principales identifiée avec leur description, leur score et le nombre de peptides ayant
La partie droite (navigation par query) représente sous la forme d'une hiérarchie de query de l'identification :
La barre d'outils placée en haut de la fenêtre permet de réaliser les opérations suivantes :