This is an old revision of the document!
Table of Contents
L'interface graphique
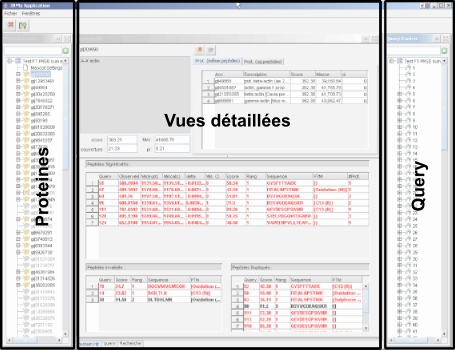
L'interface graphique d'IRMa est composé de deux panels de navigation à gauche et à droite de la fenêtre et d'un ensemble de vues détaillées au centre.
Navigation par protéines
Sur la partie gauche, ce navigateur représente sous la forme d'une hiérarchie de hits les différents élements de l'identification :
vues détaillées
La fenêtre centrale est dédiée aux vues détaillées des éléments de l'identification.
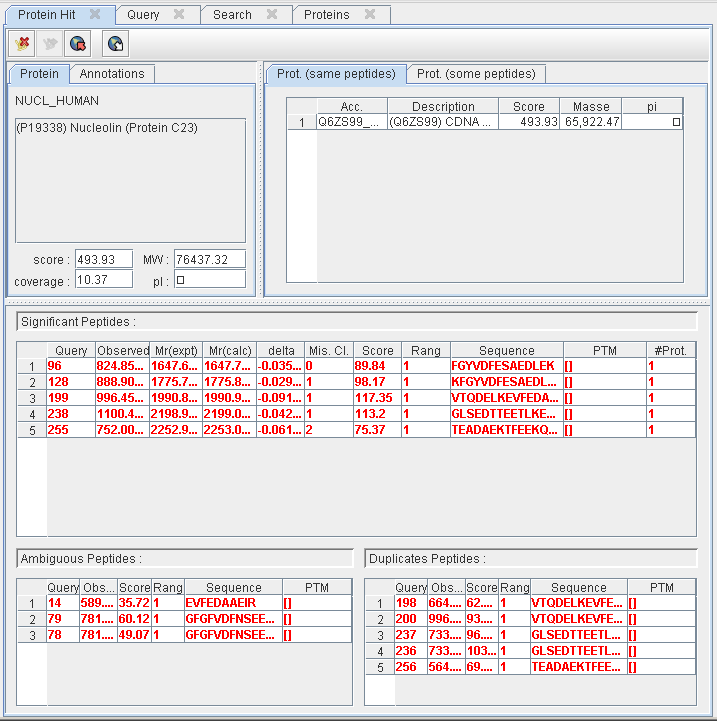
Protein Hit
La vue la plus utilisée est celle qui détaille le contenu d'un hit. Les informations suivantes sont visibles dans cette vue :
- La description de la proteine principale, son score, masse, pI, couverture, etc.
- La liste des protéines qui matchent pour un même ensemble ou pour un sous ensemble des peptides.
- Les peptides qui matchent pour ce hit.
- Une liste d'annotation obtenu pour la protéine principale
Une barre d'outil permet de :
- de supprimer le hit dans le cas où il n'y a plus de peptides significatifs pour celui-ci
- de regrouper le hit avec uin autre dans le cas où il n'y a plus de peptides significatif spécifique à ce hit.
De plus, il est possible à tout moment de changer de protéine principale en en sélectionnant une nouvelle dans la liste des protéines qui matchent pour un même ensemble de peptide. Cliquer avec le bouton droit sur la protéine désirée et choisir de la passer en protéine principale.
Au niveau des peptides, il est possible d'effectuer les opérations suivantes sur cette vue :
- Obtenir la vue Mascot d'un peptide (peptide view) via une connexion au serveur Mascot. Pour ce faire, il faut sélectionner un peptide dans l'un des tableaux et presser la barre d'espace.
- Invalider un peptide en sélectionnant un ou plusieurs peptides dans le tableau des peptides significatifs et en pressant la touche A (pour Ambigu).
Query
On retrouve ici, pour la query couramment sélectionnée, la liste des 10 peptides qui ont matchés. Pour chacun de ces peptides, il est possible de visualiser le spectre annoté et le tableau des ions fragments.
Search
Regroupe les informations sur la recherche Mascot : base utilisé, fichier soumis, taxonomie …
Proteins
Liste de toutes les protéines principales identifiée avec leur description, leur score et le nombre de peptides ayant
La partie droite (navigation par query) représente sous la forme d'une hiérarchie de query de l'identification :
La barre d'outils placée en haut de la fenêtre permet de réaliser les opérations suivantes :